Clustering
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
데이터셋 생성
sklearn패키지의 make_blobsmethod를 통해서 데이터 셋을 생성한다.
# 데이터 생성
X, y = make_blobs(n_samples = 500 , n_features = 2, centers = 4, cluster_std=1.5 , random_state =97 )
# 데이터프레임으로 재구성
target = pd.DataFrame(y)
target.columns=['Target']
data = pd.DataFrame(X)
data.columns = ['Attri_1','Attri_2']
df = pd.concat([data,target],axis=1)
df
| Attri_1 | Attri_2 | Target | |
|---|---|---|---|
| 0 | -2.634632 | -2.250270 | 1 |
| 1 | 4.532675 | -6.285143 | 2 |
| 2 | 9.182722 | 8.908761 | 3 |
| 3 | 8.472858 | 10.529204 | 0 |
| 4 | 5.582174 | 7.721625 | 0 |
| ... | ... | ... | ... |
| 495 | 9.352046 | 8.388088 | 3 |
| 496 | 8.149692 | 9.503454 | 3 |
| 497 | -0.084687 | -4.759452 | 1 |
| 498 | 5.676850 | 10.491732 | 0 |
| 499 | 8.457310 | -3.120653 | 2 |
500 rows × 3 columns
# Data Plot
col = pd.Series(df['Target']).map({0:'red',1:'blue',2:'green',3:'black'})
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.show()
# 사용할 속성 추출
feature = df[['Attri_1','Attri_2']]
feature.tail()
| Attri_1 | Attri_2 | |
|---|---|---|
| 495 | 9.352046 | 8.388088 |
| 496 | 8.149692 | 9.503454 |
| 497 | -0.084687 | -4.759452 |
| 498 | 5.676850 | 10.491732 |
| 499 | 8.457310 | -3.120653 |
K-means Clustering
실습 파일에서와 마찬가지로, 클러스터링과 시각화 및 Elbow rule을 통한 검증을 진행합니다.
시각화 진행 시, plot에 클러스터 중심의 위치를 나타냅니다.
# K-means Clustering Algorithm
from sklearn.cluster import KMeans
# 모델 생성
kmeans = KMeans(n_clusters=4,max_iter=50)
# 모델 피팅
kmeans.fit(feature)
KMeans(max_iter=50, n_clusters=4)
# 피팅 결과 생성된 클러스터 라벨
kmeans.labels_
# 각 클러스터의 클러스터 센트로이드
kmeans.cluster_centers_
array([[ 6.48539263, 9.4436022 ],
[ 5.74813556, -4.2879631 ],
[-1.00013483, -4.10072491],
[ 9.9020639 , 8.32587273]])
# SSE (Sum of Squared Distances of samples to their closest cluster center)
kmeans.inertia_
2005.1793171587194
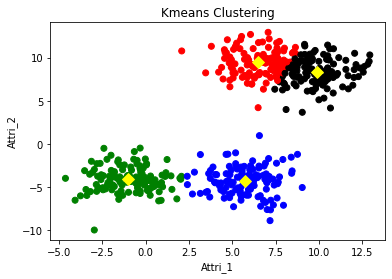
# Clustered result
col_cluster = pd.Series(kmeans.labels_).map({0:'red',1:'blue',2:'green',3:'black'})
f1 = plt.figure(1)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster)
plt.scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1], c='yellow', s=70, marker='D') # cluster centroids
plt.title('Kmeans Clustering')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
# Original data
f2 = plt.figure(2)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.title('original')
plt.show()
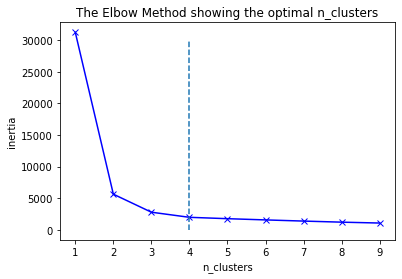
num_clusters = range(1,10)
inertias = []
for num in num_clusters:
kmeanModel = KMeans(n_clusters=num)
kmeanModel.fit(feature)
inertias.append(kmeanModel.inertia_)
plt.plot(num_clusters, inertias, 'bx-')
plt.xlabel('n_clusters')
plt.ylabel('inertia')
plt.title('The Elbow Method showing the optimal n_clusters')
plt.plot(np.linspace(4,4,6),np.linspace(-1,30000,6),'--')
plt.show()
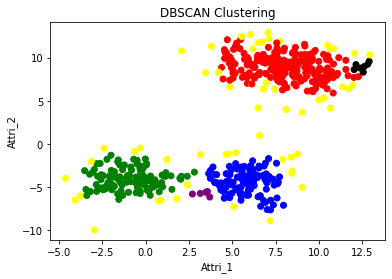
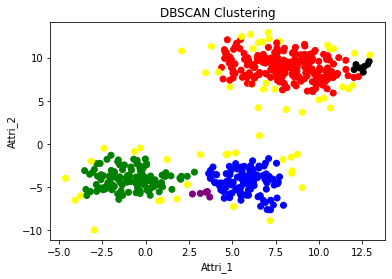
DBSCAN
실습 파일에서와 마찬가지로, 클러스터링과 시각화를 진행합니다.
# DBSCAN clustering algorithm
from sklearn.cluster import DBSCAN
# 모델 생성
dbscan = DBSCAN(eps=0.7, min_samples=5)
# 모델 피팅
dbscan.fit(feature)
DBSCAN(eps=0.7)
set(dbscan.labels_)
{-1, 0, 1, 2, 3, 4}
# Clustered result
col_cluster_1 = pd.Series(dbscan.labels_).map({-1:'yellow',0:'red',1:'blue',2:'green',3:'black',4:'purple'})
f1 = plt.figure(1)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster_1)
plt.title('DBSCAN Clustering')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
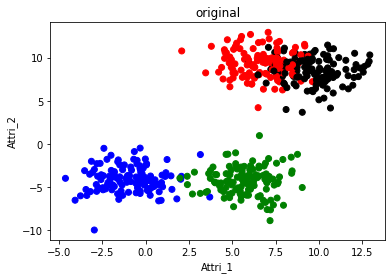
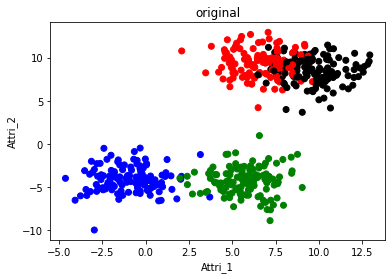
# Original data
f2 = plt.figure(2)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.title('original')
plt.show()
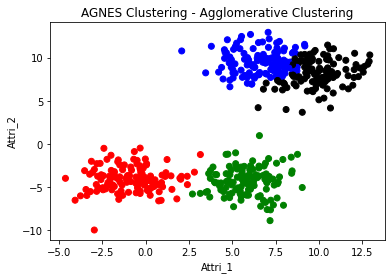
AGNES
실습 파일에서와 마찬가지로, 두 가지 클래스를 모두 이용하여 클러스터링과 시각화를 진행합니다.
후자의 경우 덴드로그램도 그려주세요!
linkage의 경우 ward 아니라 세션에서 다루었던 다른 거리 측정방식을 사용해도 좋습니다!
sklearn.cluster.AgglomerativeClustering
from sklearn.cluster import AgglomerativeClustering
# 모델 생성
agnes = AgglomerativeClustering(n_clusters=4)
# 모델 피팅
agnes.fit(feature)
AgglomerativeClustering(n_clusters=4)
# 피팅 결과 생성된 클러스터 라벨
agnes.labels_
# Clustered result
col_cluster_2 = pd.Series(agnes.labels_).map({0:'red',1:'blue',2:'green',3:'black'})
f1 = plt.figure(1)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster_2)
plt.title('AGNES Clustering - Agglomerative Clustering')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
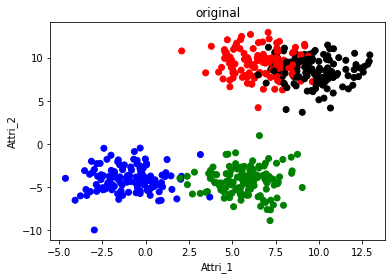
# Original data
f2 = plt.figure(2)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.title('original')
plt.show()
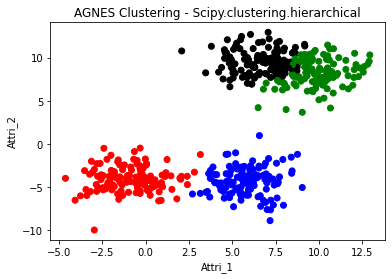
scipy.cluster.hierarchy
from scipy.cluster.hierarchy import linkage, cut_tree
# from scipy.cluster.hierarchy import ward, cut_tree
linkage_array = linkage(feature, method='ward')
# linkage_array = ward(feature)
linkage_array
array([[7.40000000e+01, 2.76000000e+02, 1.25463773e-03, 2.00000000e+00],
[3.08000000e+02, 3.40000000e+02, 1.73685772e-02, 2.00000000e+00],
[1.21000000e+02, 4.16000000e+02, 2.02554219e-02, 2.00000000e+00],
...,
[9.92000000e+02, 9.93000000e+02, 3.97321894e+01, 2.50000000e+02],
[9.94000000e+02, 9.95000000e+02, 7.53068455e+01, 2.50000000e+02],
[9.96000000e+02, 9.97000000e+02, 2.26161762e+02, 5.00000000e+02]])
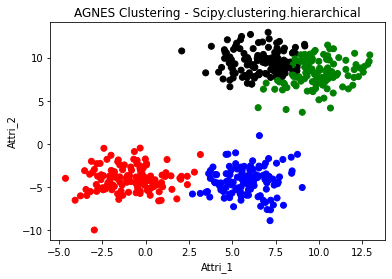
label_cluster = cut_tree(linkage_array, n_clusters=4)
label_cluster = [lab[0] for lab in label_cluster]
# Clustered result
col_cluster_2 = pd.Series(label_cluster).map({0:'red',1:'blue',2:'green',3:'black'})
f1 = plt.figure(1)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster_2)
plt.title('AGNES Clustering - Scipy.clustering.hierarchical')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
# Original data
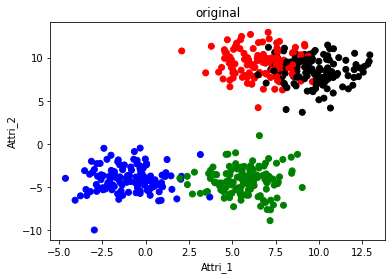
f2 = plt.figure(2)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.title('original')
plt.show()
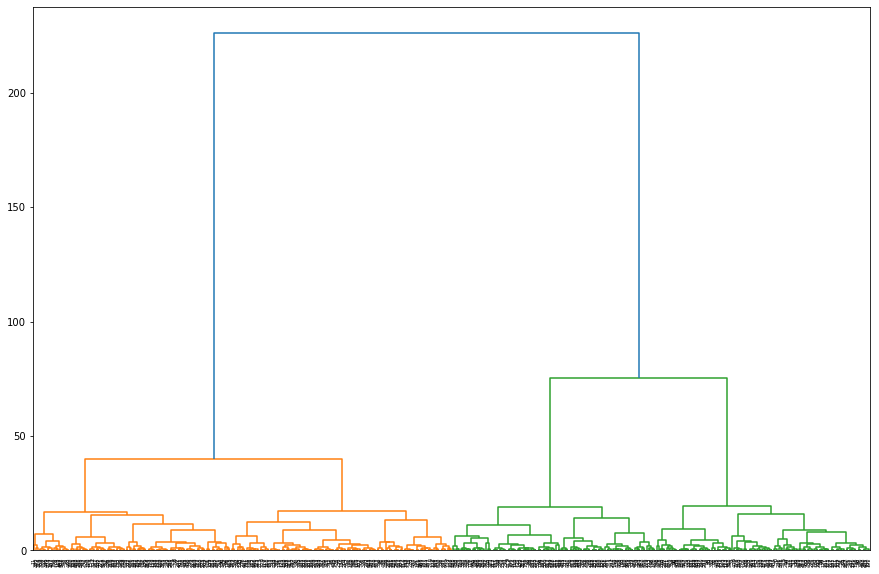
from scipy.cluster.hierarchy import dendrogram
plt.figure(figsize=(15,10))
fig = dendrogram(linkage_array)
클러스터링 결과 비교
generate된 데이터가 어떠한 클러스터링 알고리즘에서 가장 분류가 잘 되었는지, 어떤 알고리즘에서 가장 분류가 안 되었는지 보고, 그 이유를 추측해서 간단하게 써주세요!
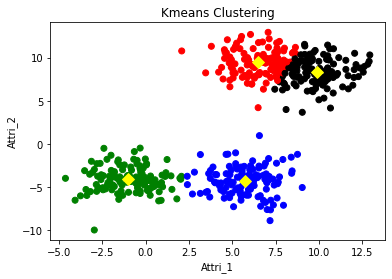
# Data Plot
col_cluster = pd.Series(kmeans.labels_).map({0:'red',1:'blue',2:'green',3:'black'})
f1 = plt.figure(1)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster)
plt.scatter(kmeans.cluster_centers_[:,0],kmeans.cluster_centers_[:,1], c='yellow', s=70, marker='D') # cluster centroids
plt.title('Kmeans Clustering')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
# Clustered result
col_cluster_1 = pd.Series(dbscan.labels_).map({-1:'yellow',0:'red',1:'blue',2:'green',3:'black',4:'purple'})
f1 = plt.figure(2)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster_1)
plt.title('DBSCAN Clustering')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
# Clustered result
col_cluster_2 = pd.Series(label_cluster).map({0:'red',1:'blue',2:'green',3:'black'})
f1 = plt.figure(3)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col_cluster_2)
plt.title('AGNES Clustering - Scipy.clustering.hierarchical')
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
# Original data
f2 = plt.figure(4)
plt.scatter(df['Attri_1'],df['Attri_2'],c=col)
plt.xlabel('Attri_1')
plt.ylabel('Attri_2')
plt.title('original')
plt.show()
결론
- AGNES가 가장 분류가 잘 되었고 DBSCAN이 가장 분류가 안 되었다.
AGNES
- AGNES의 경우 각각의 성분들이 존재하였을때 가장 가까운 것들을 천천히 합쳐 나가는 방식이다. ward를 사용하게 된다면 두 집단을 합쳤을 때 centroid가 얼마나 바뀌는가이다. 가장 적게 바뀐다면 두 가지를 합치는 방식이다.
- K-means의 경우 centroid가 그대로인 상태로 다른 점들을 포함할 것인가 아닌가를 따지는 것 이기 때문에 약간은 다르다고 할 수 있다.
- 주어진 dataset을 관찰하면, 하단에 있는 두 가지 부류는 K-mean과 AGENS가 거의 동일하게 나누었지만, 상단에 있는 두 가지 부류는 AGNES가 더 잘 분류를 했다는 것을 확인할 수 있는데, 그 이유는 다음과 같다.
- Clustering은 전쟁과 비슷하다. 도시국가들이 싸워서 합병을 하는 것인지 혹은 큰나라 두 개가 수 많은 성들을 나누어 먹는 것인지의 차이라고 생각하면 편할 것 같다.
- K-means의 경우 후자라고 할 수 있다. centroid에 가까우면 먹고 아니면 주는 형태를 취하기 때문에 위의 첫 번째 그림처럼 국경이 완벽하게 갈린 것이라고 볼 수 있다.
- AGENS의 경우 전자라고 할 수 있다. 도시국가들이 생겨나고 가장 가까운 도시국가들끼리 싸워서 이기는 나라가 점차 성장한다고 볼 수 있다. 이렇게 세력다툼을 하다가 마지막에 큰 나라 두 개가 생기는 것인데, 이 과정속에서 국경이 일직선으로 유지될 수가 없을 것이다.
- 그런데 original을 보면 black과 red가 서로 침투되어있는 것을 확인할 수 있다.
- $\therefore$ K-means보다 AGNES의 경우 상대적으로 국경이 곡선인 AGNES가 더 적합했다고 말할 수 있겠다.
DBSCAN
- DBSCAN은 commment를 남길 것이 없을 정도로 해당 데이터 셋에 대해서 최악인 이유가 명확하다.
- 상위 두 집단은 어떠한 pattern을 띠고 있지 않다. DBSCAN은 특정한 pattern을 띨 때 유용하게 쓰일 수 있는데, 오히려 겹쳐져 있는 형태를 띤다. 따라서 애초에 절대 제대로 Clustering이 될 수 없다.
이 문서는 DSL과제 template을 그래도 가져왔음.